Welche genetischen Veränderungen prädisponieren Menschen für Infektionen?
Worum geht es in diesem Forschungsprojekt?
Wir wollen verstehen, warum das Immunsystem mancher Menschen nicht in der Lage ist, Infektionen zu bekämpfen oder Entzündungen zu kontrollieren, wie es bei den meisten Menschen der Fall ist. Wir sind besonders daran interessiert, zu verstehen, wie genetische Faktoren diese Variabilität erklären könnten.
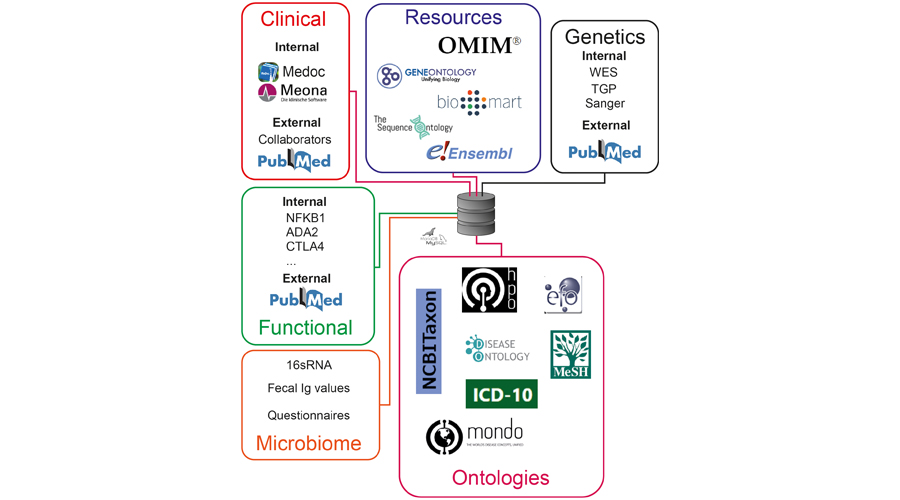
Für unsere Forschungsarbeiten nutzen wir die immunogenetische Plattform Gemma – eine Datenbank, in der Ressourcen und Forschungsdaten gespeichert werden. Mit ihr können zum Beispiel genetische Forschungsberichte automatisch erstellt sowie klinische Metadaten harmonisiert und beschleunigt erfasst werden.
Wie ist der Stand der Dinge?
Bei einem Verdacht auf eine angeborene Immunstörung kommt es oft zu einer negativen oder einer zweideutigen genetischen Diagnose und eindeutige krankheitsverursachende Mutationen können nicht nachgewiesen werden. Wir gehen davon aus, dass dies sowohl an den technischen Beschränkungen der derzeitigen Sequenzierungsplattformen liegt als auch an fehlendem Wissen.
Die Abbildung verdeutlicht das Verhältnis zwischen der Anzahl der Personen, die eine klinische Diagnose bekommen, zu der Anzahl der Personen, bei denen eine genetische Diagnose möglich ist.
Wie kommen wir da hin?
Die genetische Architektur komplexer menschlicher Krankheiten, zu denen angeborene Immunschwächen gehören, wird wahrscheinlich durch mehrere Komponenten beeinflusst. Darunter sind häufige und seltene genetische Varianten, strukturelle Varianten und Wechselwirkungen zwischen Genen und Genen sowie zwischen Gen und Umwelt. Infolgedessen müssen wir einerseits die Liste möglicher Kandidatinnen und Kandidaten erweitern, während wir andererseits unsere Annahmen über monogene oder oligogene Kausalität in vielen Situationen revidieren müssen.
Um die Technologie (z. B. die optische Genomkartierung) und das Wissen (die klinische, genetische und molekulare Expertise sowie die Daten) im Zusammenhang mit angeborenen Immunstörungen zu verbessern, richten wir derzeit eine gemeinsame „Freiburg-Hannover ImmunoGenetics Research Unit“ (FH-IGRU) ein. In dieser bündeln wir die Fachkenntnisse und Ressourcen des Instituts für Humangenetik sowie der Klinik für Rheumatologie und Immunologie der MHH mit denen der Genetik- und Genomikeinheit des CCI / IFI an der Uniklinik Freiburg.
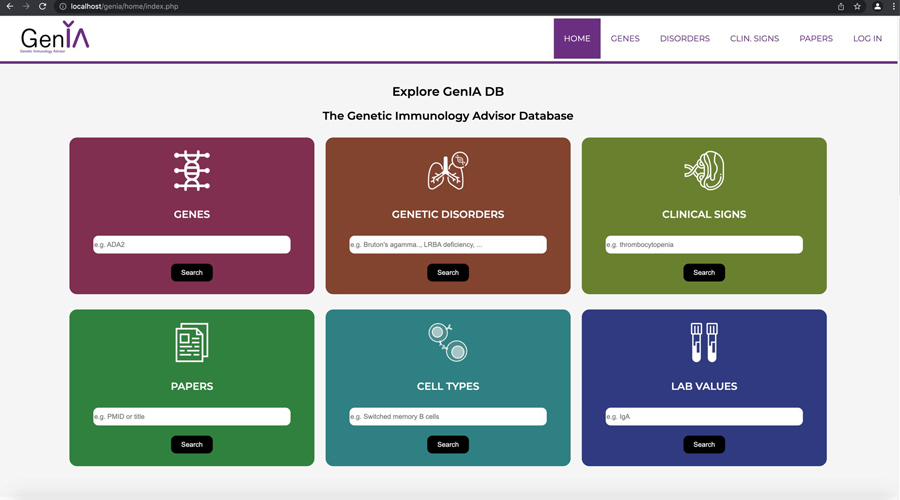
Um die große Menge an genetischen Daten effizient analysieren zu können, haben wir die Gemma-Datenbank entwickelt. Darüber hinaus wurde eine Teilmenge der öffentlich zugänglichen Daten in einem Satellitenprojekt (www.GeniaDB.net) im Internet zur Verfügung gestellt. Im Gegensatz zu anderen Datenbanken, die sich hauptsächlich aus computergestützten und verknüpften Informationen zusammensetzen, wird GeniaDB sicherstellen, dass synthetisierte und sich entwickelnde Informationen aufgenommen werden – klinische und genetische Paradigmen und Fallstricke sowie neue Konzepte und Überlegungen, die für jede Patientensituation, jedes Gen und jede Erkrankung spezifisch sind. GenIA wird auch ein praktisches Hilfsmittel für Leistungserbringer sein, das ihnen hilft, ihre genetischen und funktionellen Teststrategien, die Interpretation von Gentestergebnissen und/oder die Patientenberatung zu verbessern.