Welche Immunantwort ist besonders effizient und kann zur Entwicklung eines neuen Impfstoffes beitragen?
Worum geht es in diesem Forschungsprojekt?
Etwa 71 Millionen Menschen weltweit sind chronisch mit dem Hepatitis C Virus (HCV) infiziert und bei diesen Patienten besteht ein erhöhtes Risiko einer schweren Lebererkrankung, einschließlich eines Leberzellkarzinoms. In diesem Zusammenhang hat die WHO für 2015 von ca. 400.000 HCV-assoziierten Todesfällen berichtet. Neu entwickelte Kombinationstherapien heilen mehr als 95% der behandelten Patienten. Viele Menschen sind sich ihrer HCV-Infektion jedoch nicht bewusst und hohe Behandlungskosten schränken den Zugang zur Therapie ein. Darüber hinaus schützt eine erfolgreiche Therapie nicht vor einer erneuten Virusinfektion, die besonders in Populationen mit häufiger Virusexposition leicht auftritt. Aus diesen Gründen ist die Entwicklung eines prophylaktischen HCV-Impfstoffs eine medizinische Notwendigkeit. Tatsächlich legen Studien nahe, dass eine globale Kontrolle von HCV nur mit einem prophylaktischen HCV-Impfstoff in Kombination mit einer antiviralen Behandlung möglich ist.
Wie ist der Stand der Dinge?
Die zelluläre Immunität hat einen wichtigen Einfluss auf den klinischen Ausgang einer HCV-Infektion. Neuere Studien untermauern aber auch die Schlüsselrolle von breit neutralisierenden Antikörpern (bnAks) für die Kontrolle einer akuten oder chronischen HCV-Infektion. Ca. 20-30% der infizierten Personen eliminieren auf natürliche Weise die Infektion und Immunisierung mit rekombinanten HCV-Glykoproteinen induzieren bnAks in Tiermodellen. Diese Ergebnisse lassen hoffen, dass die Entwicklung eines prophylaktischen HCV Impfstoffs möglich ist.
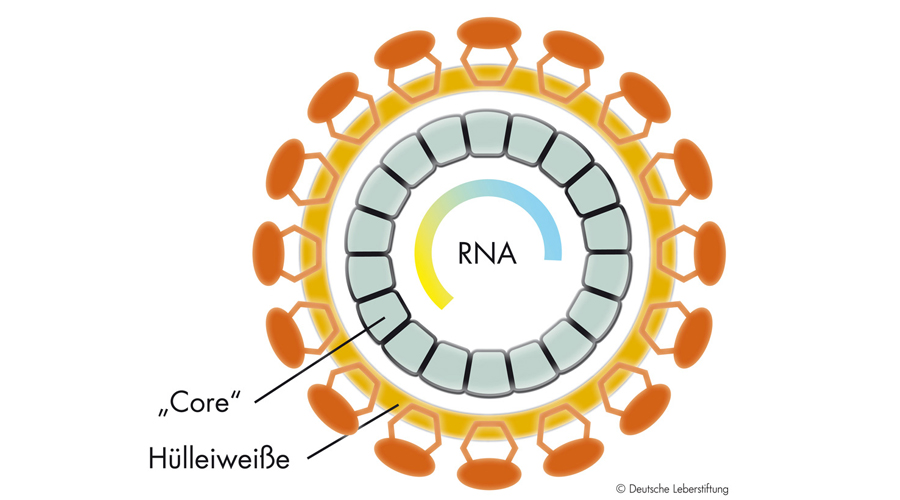
Die HCV Hüllproteine E1 und E2 sind die Hauptziele für bnAks und stehen daher im Fokus der Impfstoffforschung. HCV hat jedoch eine Reihe von Evasionsmechanismen entwickelt, die die Entwicklung von E1-E2-basierten Impfstoffen erschweren: So verringern die strukturelle Flexibilität der wichtigsten E1 und E2 Antikörperbindungsstellen („Epitope“) sowie eine reiche Ausstattung der Hüllproteine mit Zuckerresten die Immunogenität. Darüber hinaus sind die meisten Antikörper gegen hochvariable virale Epitope gerichtet, sogenannte „Köder-Epitope“ („decoy epitopes“). Diese mutieren sehr leicht und machen diese Antikörper damit unwirksam.
Wie kommen wir da hin?
Ein entscheidender Schritt in diesem Projekt wird ein großangelegtes Neutralisationsscreening sein, das die oben angesprochenen „Elite“-Neutralisierer unter den Patienten identifiziert, die in der MHH-Hepatitis-Ambulanz behandelt werden. Um diese Patientinnen und Patienten mit einer besonders effizienten Immunantwort zu identifizieren, nutzt das Projekt die Hepatitis C-Kohorte der MHH. Neben den „Elite“-Neutralisierern identifiziert dieses Screening auch Patienten mit mäßigen oder schwachen Antikörperreaktionen und ermöglicht damit vergleichende molekulare Analysen der Faktoren, die die Entwicklung leistungsfähiger Antikörper steuern. Zu diesem Zweck werden wir in diesen verschiedenen Patientengruppen mittels Einzelzellsequenzierung Profile der B-Zell Repertoires erstellen, um Merkmale zu identifizieren, die mit der Entwicklung phänotypisch unterschiedlicher Antikörperantworten korrelieren. Um potente HCV-spezifische bnAks zu identifizieren, werden wir HCV-spezifische B-Zellen von „Elite“-Neutralisierern isolieren, die B-Zell-Rezeptorgene sequenzieren und die entsprechenden Antikörper herstellen. Anschließend werden wir deren Neutralisationsstärke messen und für besonders interessante Antikörper die Immunkomplexe strukturell untersuchen. Diese Analysen werden detaillierte Strukturinformationen zu Neutralisationsepitopen und Antigenerkennung durch potente bnAks liefern. Die dadurch gewonnenen neuen Erkenntnisse werden den Weg für die Verwendung neuartiger computergestützter, strukturbasierter Verfahren für das Immunogendesign ebnen, mit dem primären Ziel, eine verbesserte Produktion epitopspezifischer – und damit breit neutralisierender – Antikörper zu erzielen.