Wie beeinflussen Darmbakterien und Infektionen die Fitness von Immunzellen?
Worum geht es in diesem Forschungsprojekt?
Infektionen sind eines der Hauptrisiken, die zur Morbidität und Mortalität von Frühgeborenen führen. Über die Immunzellreifung in Frühgeborenen und wie diese durch gesundheitsfördernde Faktoren, wie Probiotika positiv beeinflusst wird, ist nur wenig bekannt.
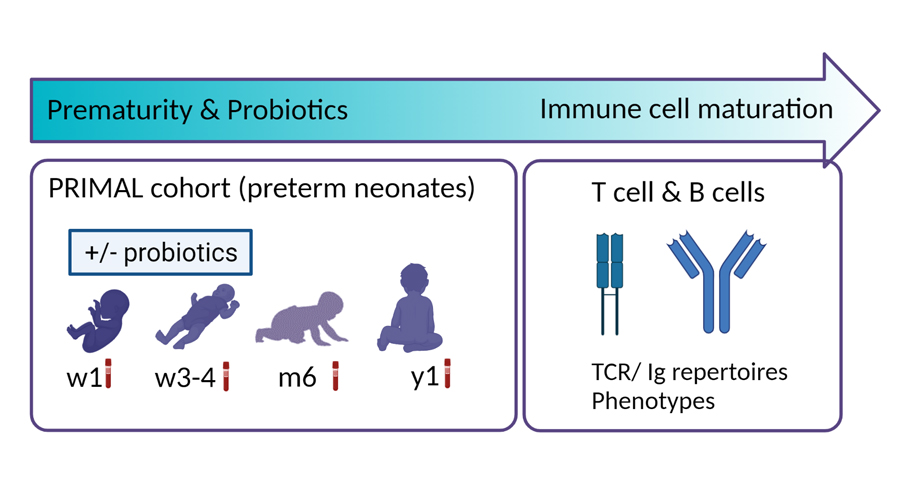
In Zusammenarbeit mit Prof. Dr. Dorothee Viemann möchten wir verstehen, welchen Einfluss die Zugabe von Probiotika auf die Immunzellreifung bei Frühgeborenen hat. Dabei konzentrieren wir uns in diesem Projekt auf Immunzellen (γδ T-Zellen, αβ T-Zellen und B-Zellen), die einen Antigenrezeptor auf ihrer Oberfläche exprimieren. Einige dieser T-Zellen und B-Zellen sind zum Zeitpunkt der Geburt voll funktionsfähig, wobei die Mehrzahl sich einer postnatalen Reifung unterzieht. Wir vermuten, dass perinatale Faktoren und die Mikrobiota das Antigen-Rezeptor-Repertoire und die Funktionalität von T- und B-Zellen stark prägen und sich auf die Anfälligkeit des Patienten für Infektionskrankheiten auswirkt.
Dieses Projekt basiert auf der PRIMAL Kohorte. Prof. Viemann leitet und koordiniert diese Studie, die dazu dient, den Effekt von Probiotika in den ersten Lebenswochen auf die Infektanfälligkeit von Frühgeborenen im ersten Lebensjahr zu verstehen. Wir möchten die Reifung von T- Zellen und B-Zellen determinieren.
Wie ist der Stand der Dinge?
Unmittelbar nach der Geburt sind Umweltfaktoren wie die Mikrobiota, sowie auftretende Infektionen wichtig für die Reifung und Prägung des Immunsystems der Kinder. Allerdings ist weitgehend unklar, welche Faktoren die Entwicklung der γδ T-Zellen, αβ T-Zellen und B-Zellen beeinflussen und wie diese sich in Bezug auf ihre Funktionalität und ihr Antigen-Rezeptor-Repertoire nach der Geburt anpassen. Zudem ist über die Unterschiede des Immunsystems zwischen Frühgeborenen und Neugeborenen wenig bekannt.
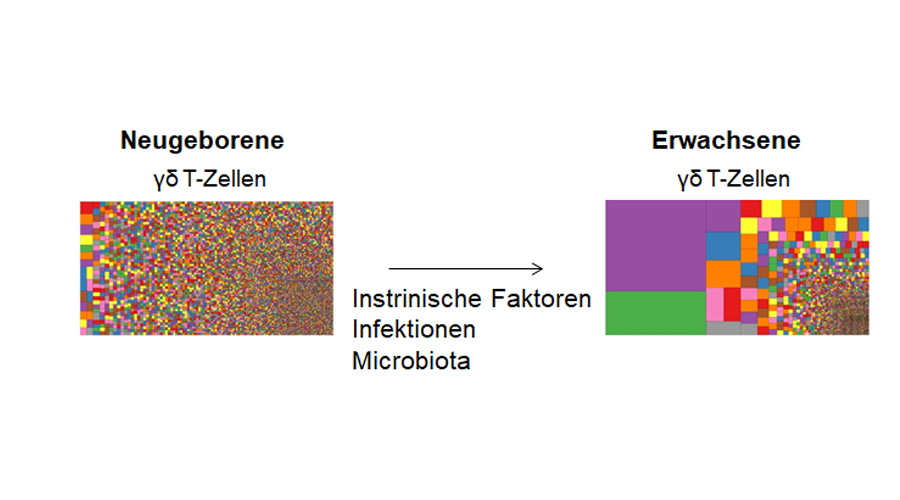
Zur Abbildung: Antigen-Rezeptor-Repertoires unterscheiden sich in Neugeborenen und Erwachsenen. Die Treemaps zeigen die Verteilung einzelner T-Zell-Klone der gamma delta T-Zellen im peripheren Blut eines Neugeborenen und eines Erwachsenen. Jedes Quadrat repräsentiert die Häufigkeit eines individuellen T-Zellklons entsprechend der Quadratgröße. Wir möchten den Einfluss von intrinsischen Faktoren, Infektionen und der Mikrobiota auf die Reifung des Antigen-Rezeptor-Repertoires im ersten Lebensjahr entschlüsseln.
Wie kommen wir da hin?
Wir haben eine mRNA-basierte Hochdurchsatzanalysetechnologie und bioinformatische Methoden zur Analyse des T-Zell-Rezeptor-Repertoires bei Neugeborenen und Erwachsenen entwickelt (Ravens et al., 2017). Erste Daten definierter Patientenkohorten weisen auf eine postnatale, extrathymische Reifung des exprimierten Antigen-Rezeptor-Repertoires hin (Ravens et al., 2017; DiLorenzo et al., 2019). Zudem hinterlassen Virusinfektionen einen individuellen Fußabdruck im Antigen-Rezeptor- Repertoire (Ravens et al., 2017; Ravens et al., 2018). Diese Ergebnisse deuten darauf hin, dass bestimmte Antigenrezeptoren als wertvolle Biomarker dienen könnten. Gleichzeitig könnten wir zeigen, dass sich bestimme T-Zellen im frühen human Thymus entwickeln und vermutlich direkt nach der Geburt in Abhängigkeit der Mikrobiota massiv expandieren (Ravens, Fichtner et al., 2020, Tan et al., 2021).
Insgesamt spiegeln diese Ergebnisse eine hohe Plastizität des frühkindlichen Immunsystems wieder und bieten neue Optionen für eine iatrogene Beeinflussung der angeborenen und adaptiven Immunität zur Förderung der Immunreife und -anpassung im frühen Leben von Neugeborenen mit hohem Risiko für infektions- und immunvermittelte Krankheiten. Im Rahmen von RESIST möchten wir gemeinsam verstehen, wie gesundheitsfördernde Faktoren, wie zum Beispiel Probiotika die funktionelle Reifung von T- und B-Zellen bei Frühgeborenen beeinflusst und wie sich das Antigen-Rezeptor-Repertoire in der frühen Kindheit adaptiert.