Welchen Einfluss hat die genetische Vielfalt auf den Bakterien-Phänotyp?
Worum geht es in diesem Forschungsprojekt?
Genetische Unterschiede sehen wir meist im Zusammenhang mit der Humangenetik. Und so ist es uns geläufig, dass zwei Menschen sich im Durchschnitt an etwa fünf Millionen Stellen im Genom unterscheiden – also nur an etwa 0,8 Prozent des gesamten Genoms. Doch eine ganz andere Dimension hat die genetische Variation bei Bakterien: Beispielsweise können sich zwei E. coli-Stämme in bis zu 60 Prozent ihres Geninhalts unterscheiden.
Dies bedeutet, dass jede Bakterienart nicht nur ein einziges Genom besitzt, sondern auch ein vielfältiges Ensemble von Genkombinationen, das als Pangenom bezeichnet wird. Wir wollen den Einfluss dieser großen genetischen Vielfalt auf bakterielle Phänotypen untersuchen.
Wie ist der Stand der Dinge?
Zu verstehen, wie Variationen im Genotyp zu Variationen im Phänotyp führen, ist ein altes Thema der Molekularbiologie. Wir erforschen, wie man die phänotypischen Folgen genetischer Varianten in Bakterien besser vorhersagen kann. Dies kann mit einer Kombination aus „vorwärts gerichteten“, mechanistischen Modellen, und „rückwärts gerichteten“ Modellen der statistischen Genomik geschehen.
Bestimmte Bakterienarten wie Escherichia coli haben so genannte offene Pangenome. Das bedeutet, dass neue Gene („akzessorische Gene“) entdeckt werden, wenn die Genome neuer Isolate sequenziert werden. Die derzeitigen Techniken der funktionellen Genomik können nicht schnell genug skaliert werden, um mit der Flut von Genen mit unbekannter Funktion Schritt zu halten, die täglich entdeckt werden.
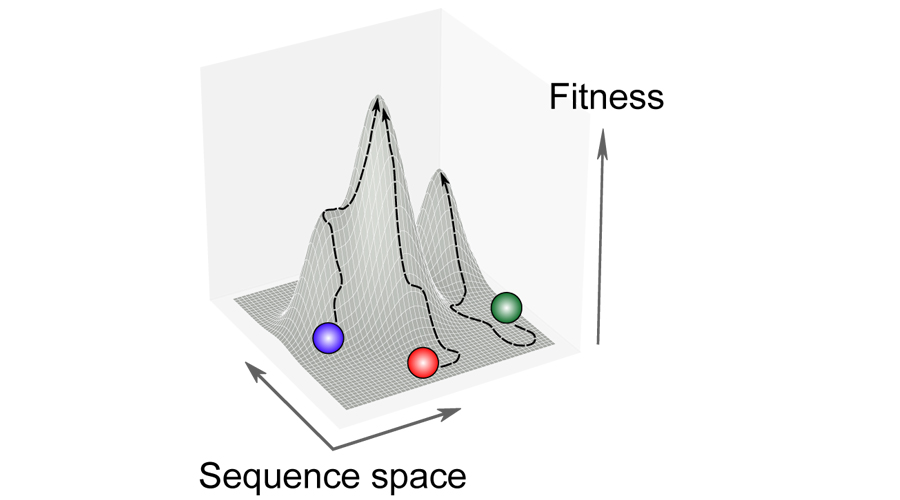
Untersuchung, wie sich verschiedene Bakterienstämme an denselben Selektionsdruck (z. B. antimikrobielle Mittel) anpassen.
Wie kommen wir da hin?
Es hat sich gezeigt, dass die genetische Variabilität zwischen den Bakterienstämmen auch die Gene betrifft, die unverzichtbar für das Wachstum und die Reproduktion sind. Wir vermuten, dass diese Unterschiede auch die Fähigkeit zur Anpassung an einen Selektionsdruck beeinflussen könnten, wie zum Beispiel den Einsatz von antimikrobiellen Mitteln. Da wir den Einfluss der bakteriellen Pangenome auf klinisch relevante Phänotypen erforschen, untersuchen wir auch, ob verschiedene genetische Hintergründe mehr oder weniger anfällig für die Entwicklung von Antibiotikaresistenzen machen.
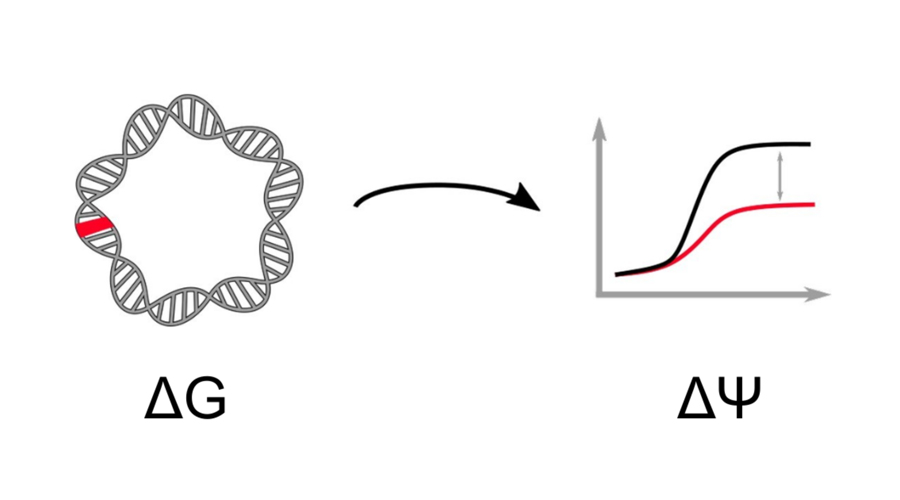
Computergestützte und experimentelle Ansätze zur Verknüpfung von genotypischer und phänotypischer Variation.