Wie kann die genaue Charakterisierung von mikrobiellen Lebensgemeinschaften zur Entwicklung von neuen Strategien gegen Biofilm-assoziierte Infektionen führen?
Worum geht es in diesem Forschungsprojekt?
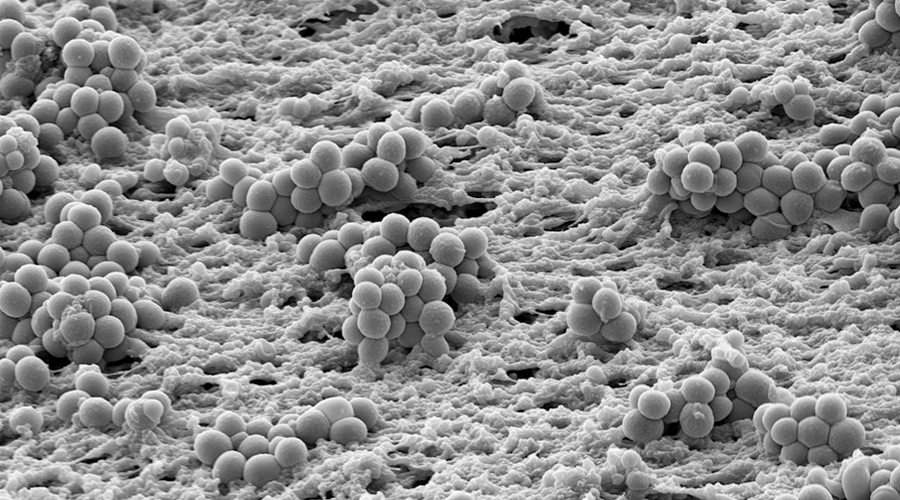
Bakterien in Biofilmen sind in eine selbst produzierte extrazelluläre Matrix eingebettet und weisen eine erhöhte Resistenz gegen widrige Umweltbedingungen auf. Im menschlichen Körper sind Biofilmbakterien für persistente Infektionen verantwortlich und widerstehen effizient Antibiotikabehandlungen und der menschlichen Immunantwort. Sobald eine bakterielle Biofilminfektion etabliert ist, wird es sehr schwierig, sie zu beseitigen – selbst wenn keine genotypische Resistenz vorliegt. Biofilm-Infektionen betreffen Millionen von Menschen, und jedes Jahr stellen chronische Infektionen bei Patientinnen und Patienten aufgrund der Biofilmbildung eine große finanzielle Belastung für die nationalen Gesundheitssysteme dar. Mit dem medizinischen Fortschritt wird immer mehr Fremdmaterial zur medizinischen Behandlung und Fremdkörperimplantate eingesetzt. Dabei sind Infektionen nach wie vor eine der größten Komplikationen. Außerdem gibt es Biofilm-Infektionen, die nicht mit Fremdkörpern in Verbindung gebracht werden, wie z.B. chronische Infektionen der Lunge von Mukoviszidose-Patientinnen und -Patienten sowie von Patientinnen und Patienten mit chronisch obstruktiven Lungenerkrankungen. Anhaltende Entzündungen und Veränderungen in der Struktur und Funktion des betroffenen Gewebes bestimmen bei diesen Betroffenen weitgehend die Morbidität und Mortalität.
Wir wollen Biomarker identifizieren, deren Vorhandensein mit der Resistenz des Pseudomonas aeruginosa-Biofilms und genetischen / metabolischen Mustern korreliert ist und die den Wechsel zur Etablierung von krankheitserregenden Biofilmen auf Implantaten charakterisieren. Dies dient der Entwicklung eines Diagnosetools für Biofilmresistenz und Krankheitserregungs-Profiling und innovativen Behandlungsstrategien, die auf Biofilm-Resistenzmechanismen abzielen.
Wie ist der Stand der Dinge?
Obwohl chronische biofilmassoziierte Infektionen ausführlich untersucht wurden, gibt es viele offene Fragen und die allgemeine Widerstandsfähigkeit von biofilmgewachsenen Bakterien ist nur unvollständig verstanden. Der erfolgreiche Einsatz von Antibiotika zur Beseitigung biofilmbedingter Infektionen hängt von unserer Fähigkeit ab, mehrere Hauptprobleme zu überwinden. Für eine gezieltere Antibiofilmtherapie sind Kenntnisse über das biofilmspezifische Resistenzprofil einzelner Bakterienisolate unerlässlich. Ebenso benötigen wir Kenntnisse darüber, wann natürliche kolonisierende Bakteriengemeinschaften in krankmachende Biofilme übergehen, z.B. auf Implantaten. Darüber hinaus müssen neue Therapieoptionen entwickelt werden, um die zweite Einschränkung der derzeitigen Behandlung zu überwinden: die allgemeine Widerstandsfähigkeit der Biofilm-Populationen.

Biofilm mit Pseudomonas aeruginosa: Lebende Zellen (grün) und tote Zellen (rot). Quelle: TWINCORE / Jann Thöming
Wie kommen wir da hin?
Unsere Forschungsgruppen verfügen über umfangreiche Expertise in der Analyse der Struktur, Zusammensetzung und mikrobiologischen Vielfalt medizinischer Biofilme. Sie haben Methoden wie DNA/RNA-Sequenzierung und maschinelle Lernmethoden angewandt, um die genomische und transkriptionelle Landschaft von infizierenden Krankheitserregern in vitro und ex vivo zu beschreiben. Im Rahmen von RESIST wollen wir die gewonnenen Erkenntnisse und Erfahrungen aus unserer Arbeit an bakteriellen Biofilmen transferieren und eine genombasierte Vorhersage bakterieller Phänotypen durch Integration komplexer OMICS-Daten etablieren, auch unter Verwendung von Machine Learning Klassifikatoren, phylogenomischem Clustering und Feature-Selection-Techniken.
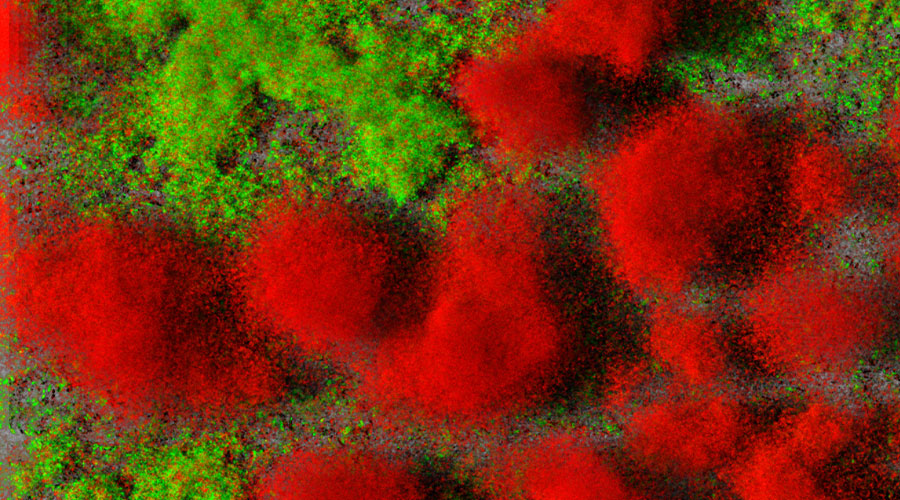
Biofilm mit Pseudomonas aeruginosa: Lebende Zellen (grün) und tote Zellen (rot). Quelle: TWINCORE / Jann Thöming