Was wirkt gegen SARS-CoV-2? Wer erkrankt trotz Impfung? Welche Rolle spielen die Gene bei der Immunantwort? Es drehen sich weiterhin zahlreiche Fragen rund um das Thema Corona. Um sie beantworten zu können, fördert das „COVID-19-Forschungsnetzwerk Niedersachsen“ (COFONI) 13 Projekte mit insgesamt rund sechs Millionen Euro. Sechs dieser Projekte leiten Wissenschaftlerinnen und Wissenschaftler, die auch in RESIST forschen beziehungsweise mit RESIST assoziiert sind. Die Projekte werden mit je bis zu 500.000 Euro unterstützt und konnten in der Zeit von Februar bis April 2022 starten:
Die Besten testen
SARS-CoV-2-Infektionen mit Antikörpern hemmen zu können – das ist das Ziel des Projekts „Aktivität humaner breit neutralisierender Antikörper in einem SARS-CoV-2 Primatenmodell“, das Prof. Schulz leitet und dessen Mit-Antragsteller auch RESIST-Forscher Prof. Krey ist. Dabei geht es dem Forschenden darum, dass die Antikörper möglichst alle SARS-CoV-2-Varianten neutralisieren können – und möglichst auch nahe verwandte tierische Coronaviren, welche menschliche Zellen infizieren und deshalb möglicherweise künftig auch auf den Menschen übertragen werden und weitere Ausbrüche verursachen könnten.
Den Forscherinnen und Forschern ist es bereits gelungen, so genannte breit neutralisierende Antikörper zu entwickeln. Diese können das ursprüngliche SARS-CoV-2-Virus, alle bisher aufgetretenen Varianten und sogar auch verwandte Betacoronaviren – beispielsweise mit SARS-CoV-2 verwandte Viren von Schuppentieren und Fledermäusen – gezielt hemmen. Das Team konnte auch schon nachweisen, dass verschiedene dieser breit neutralisierenden Antikörper Hamster vor einer Infektion sowohl mit dem ursprünglichen als auch mit der Omicron-Variante von SARS-CoV-2 schützen können. „Nun wollen wir die besten dieser Antikörper in einem Modell für nichtmenschliche Primaten testen und neue Antikörper mit einem noch breiteren Neutralisierungspotenzial zu entwickeln“, sagt Prof. Schulz. Das Projekt führen Prof. Schulz und Prof. Krey mit Prof. Pöhlmann (Deutsches Primatenzentrum, DPZ) durch.
Das Entkommen verhindern
Um hochpotente SARS-CoV-2 neutralisierende monoklonale Antikörper dreht sich auch das Projekt „Präklinische Entwicklung eines monoklonalen Antikörpers gegen SARS-CoV-2 (PREHUMAB)“, das Prof. Kalinke koordiniert. Seinem Team ist es bereits gelungen, aus Gedächtnis B-Zellen von genesenen COVID-19-Patientinnen und -Patienten hochpotente SARS-CoV-2 neutralisierende monoklonale Antikörper zu entwickeln. „Diese werden wir nun als so genannte multivalente bispezifische Reagenzien weiterentwickeln. Mit Hilfe dieser Reagenzien soll verhindert werden, dass Varianten des Virus entstehen, die der Immunabwehr entkommen können“, sagt Prof. Kalinke.
Darüber hinaus wird das Team verschiedene Wege erproben, um solche Reagenzien im Lungenepithel zur Expression zu bringen. Neben Experimenten in Mäusen und Hamstern sind auch präklinische Untersuchungen in nichthumanen Primaten geplant. So werden die Voraussetzungen für eine klinische Phase I- Untersuchung in Probanden der vielversprechendsten Antikörper erfüllt. Das Projekt führt Prof. Kalinke zusammen mit Prof. Schambach (MHH) und Prof. Pöhlmann (DPZ) durch.
Neue Bindungsstellen gesucht
Ausweichvarianten des Virus sind auch das Thema des von Prof. Čičin-Šain koordinierten Projekts „Vorhersage von Immunevasions-Mutanten (PREMUS)“.
Das Coronavirus SARS-CoV-2 benötigt die Spike-Proteine auf seiner Oberfläche, um an spezifische Rezeptoren auf der Oberfläche menschlicher Zellen binden und so in die Zellen eindringen zu können. Die entsprechende Bindungsstelle des Spike-Proteins nennt sich Rezeptorbindungsdomäne (RBD). An die RBD können Antikörper des menschlichen Immunsystems binden und so dafür sorgen, dass die Viren nicht in die Zellen eindringen können.
„PREMUS zielt darauf ab, Mutationen in dieser Bindungsstelle des Spike-Proteins zu identifizieren, die verhindern können, dass die derzeit verfügbaren monoklonalen Antikörper etwas gegen die Viren ausrichten können“, sagt Prof. Čičin-Šain. Das Forschungsteam plant, eine Bibliothek von Mutationen dieser RBDs anzulegen.
Langfristiges Ziel ist es, Bindungsstellen des Virus für Antikörper zu identifizieren, welche aber nicht in der Lage sind, zu mutieren. So soll verhindert werden, dass die Viren mithilfe von Mutationen das Binden der Antikörper verhindern und somit dem Immunsystem entfliehen können. Antragssteller des Projekts sind Prof. Čičin-Šain, Prof. Dübel und Prof. Hust (TU Braunschweig) sowie Prof. Pöhlmann (DPZ).
Die Antwort des alternden Immunsystems
Um die Langzeitfolgen einer SARS-CoV-2-Infektion bei älteren Menschen dreht sich das von Prof. Hühn koordinierte Projekt „LISE – Langfristige Immunantwort älterer Individuen gegen SARS-CoV-2“. Das Team prüft, wie stark die zelluläre und die humorale Immunantwort bei älteren Menschen nach einer SARS-CoV-2-Impfung ist, wie lange sie dauert und wie sie über die Zeit abnimmt. Zu den Antragstellern gehören auch Prof. Förster, Prof. Li, Prof. Illig, Prof. Pöhlmann (DPZ), Dr. Rösner und Prof. Werfel.
Die Forschenden untersuchen auch, ob es unterschiedliche Muster der Immunantwort gibt und welche molekularen Mechanismen diesen zugrunde liegen. „Wir widmen uns darüber hinaus dem Thema, inwieweit die Impfung Antikörper induziert hat, die in der Lage sind, aktuelle und zukünftige Varianten zu neutralisieren“, sagt Prof. Hühn. Weitere Fragen sind: Korreliert die zelluläre Zusammensetzung des Immunsystems mit der Fähigkeit, nach der Impfung eine schützende Immunantwort aufzubauen? Welche vollständig geimpften Personen haben ein hohes Risiko für eine Infektion mit aktuellen und auch zukünftigen Virusvarianten? Für LISE stellt das Niedersächsische Ministerium für Wissenschaft und Kultur Sondermittel bereit, da es sich um ein Projekte zur Erforschung von Langzeitfolgen von COVID-19 handelt.
Das Team um Prof. Hühn wendet Multi-omics-Analysen an, integriert die SARS-CoV-2-spezifischen Daten mit bestehenden Multi-omics-Daten und berücksichtigt auch persönliche Risikofaktoren. Multi-omics ist ein Analyseansatz, bei dem die Datensätze mehrere „Ome“ sind. Beispielsweise handelt es sich dabei um das Genom, alle materiellen Träger der vererbbaren Informationen einer Zelle, das Transkriptom, alle in einer Zelle vorhandenen Genabschriften, oder das Epigenom, alle chemischen Modifikationen der DNA und Histonproteine, die die Aktivität der Gene und damit die funktionellen Eigenschaften der Zelle zeitweilig festlegen.
Prognose durch Multi-omics-Daten
Auch das von Prof. Yang Li koordinierte Projekt „Aufklärung der genetischen Regulierung der Immunantwort auf eine SARS-CoV-2-Infektion mit Hilfe von Einzelzell-omics-Ansätzen“ nutzt Multi-omics-Analysen. Da das Ausmaß einer Erkrankung individuell sehr verschieden ist, geht Prof. Li der Frage nach, welche Rolle die genetische Regulierung bei der Reaktion des Immunsystems auf eine SARS-CoV-2-Infektion spielt. Mit-Antragsteller des Projekts ist Prof. Cornberg. Das Team untersucht Mechanismen der Immunantwort und des Krankheitsverlaufs auf Einzelzellebene, unter anderem anhand des Genoms, des Transkriptoms und des Epigenoms von Patientinnen und Patienten.
„Wir haben bereits in früheren Studien zeigen können, dass die menschliche Immunreaktion auf Infektionen unter anderem durch genetische Faktoren bestimmt wird und durch die Integration von Multi-omics-Daten vorhergesagt werden kann“, sagt Prof. Li. „Deswegen gehen wir davon aus, dass wir mit Hilfe von „Omics-Daten“ auch die molekularen Veränderungen bei den COVID-19-Patientinnen und -Patienten identifizieren und Reaktionen des Immunsystems auf SARS-CoV-2-Infektionen machen können.“ Die Ergebnisse sollen dazu beitragen, Vorhersagen über den Krankheitsverlauf machen und wirksame Therapien entwickeln zu können.
Das Ziel: Proteasen
Welche Rolle ein ganz bestimmtes Protein bei der SARS-CoV-2 Infektion spielt – das möchte Prof. Čičin-Šain herausfinden, in dem von ihm koordinierten Projekt PROTECT: „Die Rolle der TMPRSS2-Proteolyse bei der Ausbreitung von SARS-CoV-2 in Zellen des Respirationstrakts und in vivo“. Antragssteller sind außer ihm Prof. Pöhlmann (DPZ), Prof. Ulrich (MHH) und Prof. Braun (Fraunhofer ITEM).
Mit seinem Team konzentriert er sich dabei auf die Transmembran-Serinprotease 2 (TMPRSS2). Dies ist ein Zelloberflächenprotein, welches im Atemtrakt vorkommt und an der Spaltung von Peptidbindungen bestimmter Proteine beteiligt ist. Hier ist die zentrale Frage: Welche Rolle spielt dieses Protein beim Eintritt des SARS-CoV-2-Virus in die Zelle sowie bei der Infektion?
Das Team nimmt an, dass diese zelluläre Protease ein molekulares Ziel sein könnte für therapeutische Strategien gegen die COVID-19-Erkrankung. „Dies ist besonders seit dem Auftreten der Omicron-Variante interessant, die in vitro gezeigt hat, dass die TMPRSS2-Protease weniger eindeutig benötigt wird“, sagt Prof. Čičin-Šain. Die an diesem Projekt beteiligten Labor-Teams werden die SARS-CoV-2-Infektion sowohl in menschlichen Gewebekulturen als auch in Lungenschnitten und in Nager- und Primatenmodellen untersuchen.
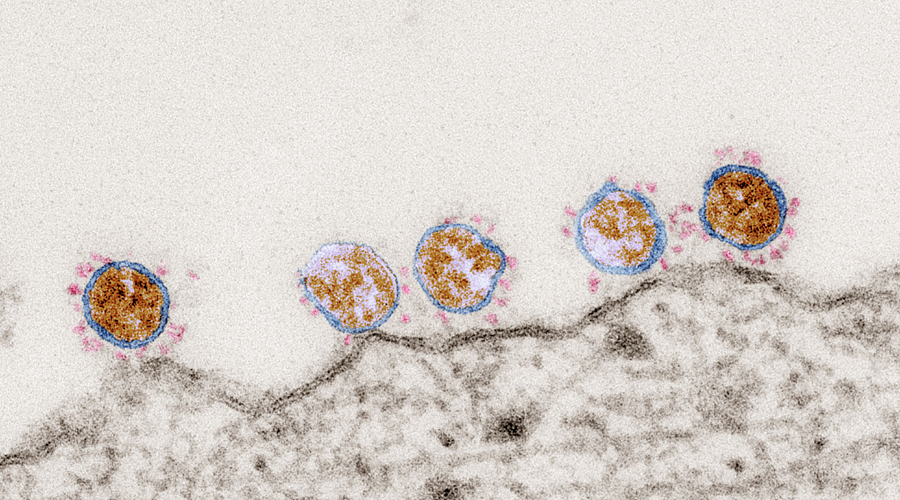
Die Abbildung zeigt SARS-Coronavirus-2-Partikel auf der Oberfläche einer Zelle (elektronenmikroskopische Aufnahme, Ultradünnschnitt).
Copyright: Tobias Hoffmann, Robert Koch-Institut (RKI), 2020